Cultivo bidimensional de MSC derivadas del cordón umbilical
Las MSC del cordón umbilical (ucMSC) se obtuvieron del Anthony Nolan Cord Bank, se cortaron en trozos pequeños y se sembraron en medio basal (es decir, αMEM + 1% v/v de penicilina-estreptomicina) que contenía 5% (v/v) de plaquetas humanas. lisado a 37 °C, 5% CO2. Después de retirar las células no adherentes y lavar con PBS, se repuso el medio y las células se cultivaron hasta alcanzar una confluencia del 70 al 90%. Luego se realizó tripsinización para una mayor caracterización y paso de células. Las ucMSC se caracterizaron por la positividad de CD90, 105, CD106 y CD73, el antígeno leucocitario humano de clase I y la falta de expresión de CD14, CD31 y CD45. Primeros pasajes de 1×106 Las ucMSC (pasos 2 a 5) se cultivaron continuamente en un filtro de 175 cm2 matraz (Corning) en medio basal + 5% (v/v) de lisado de plaquetas humanas. Cuando las células alcanzaron una confluencia del 70% al 80%, los medios se reemplazaron con medio basal antes de recolectar CCM al día siguiente para el aislamiento de EV y el subcultivo mediante un protocolo de tripsinización convencional.
Cultura tridimensional de ucMSC.
Se utilizó una placa de cultivo de micropocillos Aggrewell400 para generar esferoides ucMSC siguiendo un protocolo previamente establecido.15. Brevemente, se agregaron 500 µl de solución de enjuague antiadherente a cada pocillo antes de la centrifugación a 2,000g durante 2 min para eliminar las burbujas y la incubación durante 30 min-2 h a temperatura ambiente. Luego se lavó el Aggrewell con 500 µl de PBS por pocillo, seguido de la adición de 500 µl de medio basal y se centrifugó a 2,000g durante 2 min para eliminar las burbujas. Luego, el medio basal se reemplazó con la suspensión celular obtenida de la tripsinización 2D (preparada a una densidad de 1.2 × 105 células por 500 µl por pocillo en el medio basal suplementado con 20% v/v de reemplazo de suero KO según las instrucciones del fabricante). Luego la placa se centrifugó a 200g durante 5 minutos para la agregación celular en el fondo de cada micropocillo y se mantuvo sin molestias en la incubadora. La CCM se cosechó el día 3 y cada 2 a 3 días para el aislamiento de EV. Se repuso medio nuevo después de la recolección del medio para mantener el cultivo esférico de ucMSC durante 12 días.
Aislamiento de vehículos eléctricos
El aislamiento de EV se realizó como se describió en detalle anteriormente.46. Se llenaron tubos de ultracentrífuga de policarbonato ultraclaro (número de catálogo 355631, Beckman Coulter) con 22.5 ml de CCM filtrado (preparado filtrando CCM usando un filtro de jeringa de 0.22 µm). Luego, 3 ml de solución de sacarosa al 25% p/p (preparada en D2O) se colocó en capas lentamente debajo del CCM usando una pipeta Pasteur de vidrio. La centrifugación utilizando un rotor oscilante (SW32 Ti, Beckman Coulter) se realizó a 100,000g durante 1.5 h a 4 °C. Luego se recogió la solución de sacarosa (2 ml por tubo) y se sometió a una etapa de lavado para la purificación de EV añadiendo a tubos de ultracentrífuga de policarbonato precargados (número de catálogo 355618; Beckman Coulter) con 20 ml de PBS filtrado antes de la ultracentrifugación a 100,000g durante 1.5 h a 4 ° C utilizando un rotor de ángulo fijo (70 Ti, Beckman Coulter). Se descartó el sobrenadante y el sedimento de EV obtenido se resuspendió en PBS filtrado. Los vehículos eléctricos se mantuvieron a 4 °C durante 1 semana de almacenamiento y a -80 °C para almacenamiento a largo plazo.
Detección de marcadores EV mediante dot-blot
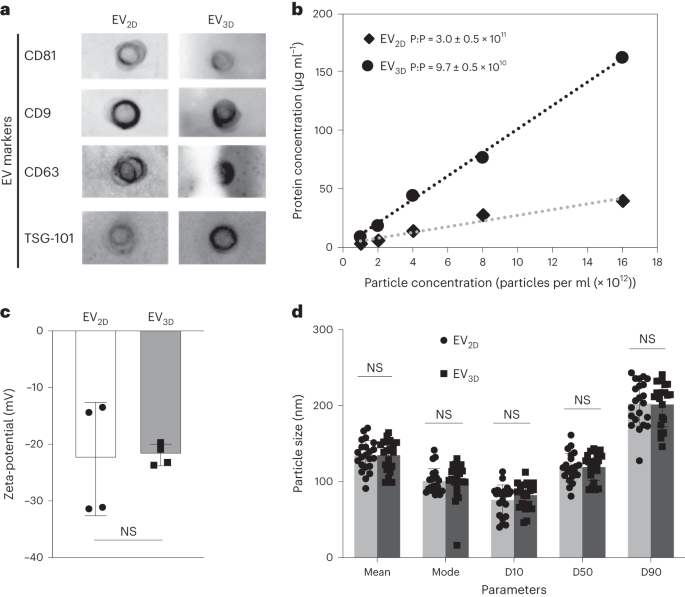
El análisis se realizó siguiendo un protocolo previamente publicado.15. Primero, 50 µl de EV a una concentración de 5 × 1010 Se detectaron partículas por ml en una membrana de nitrocelulosa (una membrana por marcador) (Bio-Rad). La membrana se secó con gas nitrógeno antes de la etapa de bloqueo usando tampón de bloqueo (es decir, leche desnatada al 3 % preparada en solución salina tamponada con Tris con Tween 0.1 al 20 % (TBS-T)) durante 1 h a temperatura ambiente. Los anticuerpos primarios (CD9, CD63, CD81 y TSG101) se agregaron individualmente en tampón de bloqueo nuevo seguido de incubación durante la noche a 4 °C (dilución 1:1,000). Luego la membrana se lavó tres veces en TBS-T, 5 minutos cada lavado. Luego se añadió el anticuerpo secundario conjugado con peroxidasa de rábano picante al tampón de bloqueo nuevo antes de una incubación adicional durante 1 hora a temperatura ambiente (1:20,000 para anti-ratón y 1:1,000 para anti-conejo). La membrana se lavó como se mencionó anteriormente y las señales se desarrollaron mediante la adición de sustrato (SuperSignal West Femto Maximum Sensitivity Substrate), seguido de imágenes utilizando el sistema Gel Doc (Bio-Rad) y análisis mediante el software Image Lab (Bio-Rad).
Ensayo de proteínas para determinar la concentración de proteínas de vehículos eléctricos aislados.
Las concentraciones de proteínas se determinaron mediante ensayo microBCA en una placa de 96 pocillos siguiendo las instrucciones del fabricante adaptadas para vehículos eléctricos. Brevemente, muestras de EV (concentración mínima, 5 × 1010 partículas por ml) se diluyeron 1:1 en PBS. Se añadió una mezcla de reactivos MicroBCA (preparada según las instrucciones del fabricante) para duplicar 40 µl de muestras diluidas (50 µl por pocillo), seguido de una incubación a 37 °C durante 1 h. La medición se comparó con BSA diluida en serie como estándar (preparada por duplicado). La absorbancia se leyó a 562 nm utilizando un lector de placas FLUOStar Omega (BMG LabTech). Se utilizó el software MARS v.2.40 (BMG LabTech) para el análisis extrapolando los valores de la curva estándar utilizando una ecuación polinómica de tercer orden, con r2 > 0.999 para cada ensayo.
NTA
NTA midió el tamaño y la concentración de los vehículos eléctricos utilizando un Nanosight LM10 (Malvern Instruments) equipado con un láser de 488 nm. El nivel de la cámara se ajustó automáticamente y el umbral de detección del análisis se estableció en 3–4. Las muestras de EV se diluyeron en PBS filtrado para obtener concentraciones óptimas (20 a 80 partículas por cuadro). Se realizaron cuatro grabaciones de vídeo con una duración de 40 s para cada preparación de EV. Se utilizó el software Nanosight NTA 3.2 (Malvern Instruments) para analizar el vídeo grabado.
Potencial zeta
La movilidad electroforética dinámica de los vehículos eléctricos y los HC-EV se midió con un software Malvern Zetasizer Nano ZS y Zetasizer v.7.12 (Malvern Instruments). Antes de las mediciones, muestras de EV (concentración mínima, 1 × 1011 partículas por ml) se diluyeron 1:50 en agua desionizada. Las mediciones se realizaron a 25 °C para cada triplicado experimental.
Preparación de corona proteica.
El recubrimiento de corona de proteína se realizó siguiendo un protocolo publicado.47 con modificaciones. Para eliminar los vehículos eléctricos y el suero proteico agregado, el FBS se sometió a ultracentrifugación a 100,000g durante 18 h a 4 °C. El sobrenadante se recogió y se filtró a través de filtros de 0.22 µm (EV-D FBS). vehículos eléctricos (~7 × 1011 partículas, equivalente a 0.05 m2) se prepararon en un volumen total de 300 µl de PBS estéril suplementado con solución de penicilina-estreptomicina al 1% (sPBS-PS) en tubos Eppendorf de 1.5 ml antes de incubar con 300 µl de EV-D FBS durante 1 h a 37 °C, 300 rpm. Tras la incubación, la mezcla se transfirió a un tubo de ultracentrífuga (número de catálogo 343778, Beckman Coulter). Las muestras fueron ultracentrifugadas a 100,000g durante 1 h a 4 °C. El sobrenadante se eliminó cuidadosamente sin alterar el sedimento. El sedimento se lavó adicionalmente con 1 ml de sPBS-PS dos veces, siguiendo las condiciones mencionadas anteriormente. En estudios de desorción de corona, en el lavado final, el sedimento (HC-EV) se dispersó en 100 µl de SDS al 2% p/v recién preparado, solución de Tris-HCl 62.5 mM, seguido de incubación a 95 °C durante 5 minutos para desorber. las proteínas que constituyen el HC. Luego se separó el HC de los EV mediante ultracentrifugación como se indicó anteriormente. El sobrenadante que contenía HC se recogió y se sometió a determinación de proteínas mediante ensayo microBCA. Las soluciones de HC se almacenaron a -80 ° C antes de realizar más análisis.
SDS-PÁGINA
Se mezclaron muestras de EV, HC-EV, EV-D FBS, KO y HC (15 µg) con tampón de muestra LDS y tampón RIPA (mezclados con un cóctel de inhibidor de proteasa 1:100) y se incubaron durante 10 minutos a 70 °C. Luego, las muestras se aplicaron en un gel de proteína Bis-Tris NuPAGE 4–12% y se procesaron durante 30 minutos a 100 mV y 1 hora más a 150 mV. Para la tinción con plata, el gel se fijó en 100 ml de solución fijadora (50% metanol, 10% ácido acético, 50 µl de formaldehído) durante la noche, seguido de lavado con 50% etanol tres veces. Luego, el gel se sensibilizó mediante hiposolución (tiosulfato de sodio al 0.02 % p/v) durante 1 min, seguido de impregnación del gel con nitrato de plata al 0.2 % p/v durante 30 min. Luego se lavó el gel con agua desionizada tres veces y se sometió a desarrollo de bandas usando 100 ml de solución de revelado (6 g de carbonato de sodio, 2 ml de hiposolución y 50 µl de formaldehído). Una vez revelada, la reacción se detuvo con ácido acético al 5% v/v. Se tomó una fotografía digital del gel sobre un fondo blanco.
Digestión de la proteína corona para análisis de espectrometría de masas.
El análisis proteómico de las proteínas FBS, EV y sus HC mediante LC-MS se realizó utilizando el método descrito por Schottler et al.47. Brevemente, se eliminó el SDS de las muestras mediante columnas de eliminación de detergente Pierce y se precipitaron 25 µg de cada muestra de proteína mediante el kit de precipitación de proteínas ProteoExtract siguiendo el manual del fabricante. Los sedimentos de proteína resultantes se resuspendieron en RapiGest SF al 0.1 % en bicarbonato de amonio 50 mM y se incubaron durante 15 minutos a 80 °C. Se añadió ditiotreitol (concentración final, 5 mM) para reducir las proteínas y la mezcla se incubó a 56 °C durante 45 min. Luego se añadió yodoacetamida (concentración final, 15 mM) y la mezcla se incubó adicionalmente en la oscuridad durante 1 hora a temperatura ambiente. Las muestras de proteínas se digirieron durante 18 h a 37 °C con tripsina con una relación enzima:proteína de 1:50 (p/p). La reacción se detuvo mediante la adición de 2 µl de ácido clorhídrico. Los péptidos digeridos se diluyeron con ácido fórmico al 0.1% v/v en agua de calidad UPLC-MS y se agregaron 50 fmol µl.-1 Hi3 Escherichia coli estándar para la cuantificación absoluta.
Captación celular in vitro de EV y HC-EV.
EV2D y EV3D fueron etiquetados según nuestro protocolo publicado48. La concentración en partículas por ml se obtuvo mediante NTA como se mencionó anteriormente. La intensidad de fluorescencia de los vehículos eléctricos recién preparados (100 µl) se midió utilizando un lector de placas FLUOStar Optima (BMG Labtech), con longitudes de onda de excitación y emisión de 485 nm y 520 nm, respectivamente. Cuando hay una diferencia en la eficiencia del etiquetado entre los lotes o tipos de vehículos eléctricos, las muestras se mezclaron con vehículos eléctricos no etiquetados para que la intensidad de la fluorescencia y el número de partículas sean comparables para las diferentes muestras. Se sembraron células fagocíticas, es decir, J774 y macrófagos derivados de monocitos humanos, y células no fagocíticas, es decir, HepG2, a una densidad de 1.5 × 105 células por pocillo en placas de 24 pocillos (Corning). Después de 24 h, los medios se cambiaron a medios libres de suero y las células se trataron con EV marcado.2D, HC-EV2D, VE3D y HC-EV3D de valores unitarios de fluorescencia comparables y una dosis de 2 × 109 partículas y se incubaron durante 1 h, 4 h y 24 h. Para evaluar la contribución del receptor de unión a albúmina en la mediación de la internalización de EV, se trataron células HepG2 con BSA (12.5 mg ml-1), seguido de la incorporación de vehículos eléctricos con la etiqueta DiD3D, durante 24 h. La citometría de flujo se realizó en un BD FACSCalibur utilizando el software BD FACStation v.6.0 (BD Biosciences) en las células separadas con tripsina-EDTA al 0.05%, se lavaron y resuspendieron en 200 µl de PBS. Todas las muestras se analizaron utilizando FlowJo v.10.7.2 (TreeStar/BD Bioscience). Las células estaban cerradas por su dispersión frontal y lateral. La absorción de vehículos eléctricos fue evaluada por el MFI de Alexa Fluor 488. El cambio en el MFI se calculó con respecto al aumento relativo del MFI con respecto a los controles no tratados.
Animales
Todos los experimentos con animales se realizaron de conformidad con la Ley de Animales (Procedimientos Científicos) del Reino Unido de 1986 y el Código de Prácticas del Ministerio del Interior del Reino Unido para el alojamiento y cuidado de animales utilizados en procedimientos científicos (Ministerio del Interior 1989). La experimentación in vivo cumplió con la licencia del proyecto aprobada por el organismo de revisión ética y bienestar animal del King's College de Londres (AWERB) y el Ministerio del Interior del Reino Unido (PBE6EB195 y PP8950634). La investigación animal y el cuidado veterinario se realizaron en el edificio Franklin-Wilkins, King's College London según el protocolo aprobado para este estudio por un Oficial designado de capacitación y competencia (Julie Keeble) y un Oficial designado de cuidado y bienestar animal (Jayne Morgan). Se obtuvieron ratones hembra CD-1 (~ 25–35 g, 5 semanas de edad) de Charles River para un estudio de biodistribución in vivo. Se obtuvieron ratones C57BL/6 machos y hembras (18–25 g, 6–8 semanas de edad) de Charles River para un estudio de captación celular in vivo. Todos los ratones se alojaron en un ciclo de 12 h de luz/12 h de oscuridad con la temperatura mantenida entre 65 y 75 °F (~18–23 °C) y ~50% de humedad. En el estudio se utilizaron ambos sexos, cumpliendo con las recomendaciones publicadas recientemente por el Consejo de Investigación Médica del Reino Unido para realizar investigaciones en animales.
Biodistribución in vivo de vehículos eléctricos mediante imágenes ópticas de fluorescencia
La CCM filtrada se incubó con trazador lipófilo fluorescente DiR 1 µM (yoduro de 1,1-dioctadecil-3,3,3,3-tetrametilindotricarbocianina) a temperatura ambiente durante 1 h con agitación antes del aislamiento de EV mediante cojín de sacarosa más ultracentrifugación como se describió anteriormente para eliminar el tinte no ligado. El experimento se realizó en ratones CD1, divididos aleatoriamente en grupos. Vehículo eléctrico recién purificado con la etiqueta DiR2D y EV3D (2 × 1011 partículas en 200 µl) se inyectaron por vía intravenosa a través de la vena de la cola. A los ratones de control se les inyectó PBS únicamente o PBS que contenía DiR. Se tomaron imágenes de ratones vivos sedados con isoflurano utilizando el sistema IVIS Lumina III (excitación, 740 nm; emisión, 840 nm) a las 1, 4 y 24 h después de la inyección intravenosa antes de matar a los animales. Se recolectaron los órganos principales (cerebro, corazón, pulmón, hígado, estómago, bazo, riñón e intestino) para obtener imágenes de fluorescencia ex vivo. La intensidad de fluorescencia en cada órgano (eficiencia radiante total) se obtuvo utilizando el software Living Image v.4.7.3 (PerkinElmer) para determinar la biodistribución en órganos de EV marcado con DiR.2D y EV3D dibujando las regiones de interés (ROI) de cada órgano. Luego, los valores se normalizaron al peso de los órganos (eficiencia radiante total por g). Para el estudio de aclaramiento renal, tras la inyección de EV marcados con DiR, los ratones se alojaron individualmente en una jaula metabólica circular estándar (Nalgene Nunc) durante 24 h, y la orina se recogió en un tubo Nalgene en el fondo de un sistema de embudo para una mayor fluorescencia. determinación de la intensidad utilizando un sistema IVIS Lumina III y el software Living Image v.4.7.3 (PerkinElmer) dibujando ROI que cubren un recipiente de orina para obtener una eficiencia radiante total por muestra de orina.
Captación celular in vivo de vehículos eléctricos en subpoblaciones de hígado
EV2D y EV3D se marcaron con DiD (DiIC18 (5); 1,1′-dioctadecil-3,3,3′,3′-tetrametilindodicarbocianina, sal de 4-clorobencenosulfonato) utilizando el mismo procedimiento que para el marcaje DiR en los estudios in vivo. El experimento se realizó en ratones C57BL/6, divididos aleatoriamente en grupos. Vehículo eléctrico con etiqueta DiD recién purificado2D y EV3D (2 × 1011 partículas en 200 µl) se inyectaron por vía intravenosa a través de la vena de la cola. A los ratones de control se les inyectó PBS únicamente o PBS que contenía DiD. Para el estudio de bloqueo del receptor de albúmina, se utilizaron 100 µl de BSA diluidos en PBS (10 mg ml-1, dosis seleccionada según la literatura49,50,51,52) se inyectó 5 minutos antes de la EV3D inyección (2 × 1011 partículas en 100 µl). Para el estudio de la internalización de vehículos eléctricos recubiertos con albúmina, para preparar vehículos eléctricos recubiertos con albúmina y marcados con DiD2D, se cultivaron ucMSC 2D (70–80 % de confluencia) en medio basal suplementado con 2.5 mg ml-1 BSA, seguida de recolección de medio después de 24 h de cultivo, etiquetado DiD y aislamiento de EV como se mencionó anteriormente. EV marcado con DiD recubierto de albúmina2D Luego se inyectaron por vía intravenosa (2 × 1011 partículas en 200 µl). Veinticuatro horas después, los ratones fueron anestesiados con fenobarbital y sometidos a disección de piel en la línea media ventral para abrir la cavidad peritoneal para la perfusión hepática siguiendo el protocolo publicado previamente con modificaciones.53. Se perfundieron a los ratones 30 ml de HBSS-EGTA calentados a 41 °C a través de la vena cava inferior usando una bomba peristáltica (SciQ 300, Watson Marlow) y un equipo de infusión con alas de 27G × 0.38” × 12” (BD Valu-Set) a una velocidad de 15 rpm La vena porta hepática se cortó después de que el hígado se hinchó y decoloró, después de lo cual la velocidad se ajustó a 20 rpm. Luego se perfundieron a los ratones con 25 ml de HBSS-CaCl que contenía colagenasa.2 (concentración, 1 mg ml-1) se calentó a 41 °C, a una velocidad de 15 rpm. El hígado se transfirió a una placa de Petri que contenía HBSS-CaCl frío.2 buffer. Las células hepáticas se liberaron rompiendo suavemente la cápsula de Glisson y agitando el hígado. Una vez obtenida una suspensión celular homogénea, las células se filtraron a través de un colador de 70 µm (Corning). Los hepatocitos se separaron mediante centrifugación durante 3 min a 50g a 4 °C con freno bajo, tres vueltas. Después de cada giro, el sedimento se resuspendió en 30 ml de HBSS-CaCl frío.2, y el sobrenadante se recogió para un posterior fraccionamiento de NPC. La fracción de NPC se sedimentó a 650g a 4 °C y se sometió a incubación con tampón de lisis de RBC durante 5 minutos en hielo, seguido de la adición de 20 ml de PBS para detener la reacción y una centrifugación adicional a 650g a 4 ° C para sedimentar la fracción de NPC purificada. La identificación de cada subpoblación se realizó en base a la expresión del marcador utilizando anticuerpos marcados fluorescentemente para análisis mediante citometría de flujo (BD FACSCelesta, operado por el software BD FACSDiva v.9.2, BD Biosciences) y el software FlowJo v.10.7.2 (TreeStar/BD Bioscience ). Los hepatocitos se tiñeron con anticuerpo anti-ratón ASGPR1 (1:200)54,55,56 y CD9 antihumano mediante tinción intracelular usando Triton-X 0.1 al 100% en PBS. Las células de Kupffer se tiñeron con CD45, F4-80 y CD11b anti-ratón (1:200, cada uno)57,58,59. Las células endoteliales se tiñeron con CD45 anti-ratón (1:200), CD31 (1:200) y CD146 (1:100).60,61,62. Las células estrelladas se tiñeron con CD45 anti-ratón (1:200), GFAP (1:50) y se detectaron utilizando un filtro 450/50 y un láser violeta de 405 nm.63,64,65. Todas las fracciones se tiñeron con Zombie Aqua para determinar la viabilidad de las células. La tinción se realizó durante 30 min a temperatura ambiente. La frecuencia de las células madre que absorben vehículos eléctricos se definió comparando la población de células DiD positivas con el grupo de control (PBS). Las MFI de la señal DiD expresadas por cada tipo de célula se utilizaron para evaluar la cantidad de absorción de vehículos eléctricos.
análisis estadístico
Los análisis estadísticos de los datos se realizaron utilizando Prism 9.4.1 (software GraphPad) mediante el uso de análisis de varianza unidireccional (ANOVA) con la prueba post hoc de Tukey para todos. P valores (*P < 0.05, **P <0.01, ***P < 0.001, ****P <0.0001; P > 0.05 no fue significativo). Todos los resultados se expresan como media ± sd. Se asumió que la distribución de los datos era normal, pero esto no se probó formalmente. No se utilizaron métodos estadísticos para predeterminar los tamaños de muestra, pero los tamaños de muestra se eligieron en base a experimentos previos realizados de manera similar por nuestro grupo con efectos estadísticamente significativos comprobados.10. Todos los gráficos se realizaron en Prism 9.4.1. Se utilizó MATLAB 9.11 para generar mapas de calor de datos. La recopilación y el análisis de datos no se realizaron a ciegas con respecto a las condiciones de los experimentos. Todos los puntos de datos se incluyeron para los análisis.
Resumen de informes
Más información sobre el diseño de la investigación está disponible en el Resumen de informes de la cartera de naturaleza vinculado a este artículo.
- Distribución de relaciones públicas y contenido potenciado por SEO. Consiga amplificado hoy.
- PlatoData.Network Vertical Generativo Ai. Empodérate. Accede Aquí.
- PlatoAiStream. Inteligencia Web3. Conocimiento amplificado. Accede Aquí.
- PlatoESG. Carbón, tecnología limpia, Energía, Ambiente, Solar, Gestión de residuos. Accede Aquí.
- PlatoSalud. Inteligencia en Biotecnología y Ensayos Clínicos. Accede Aquí.
- Fuente: https://www.nature.com/articles/s41565-023-01585-y
- :es
- :no
- ][pag
- $ UP
- 000
- 001
- 01
- 05
- 1
- 10
- 100
- 11
- 12
- 15%
- 150
- 16
- 175
- 1996
- 2%
- 20
- 200
- 2000
- 2010
- 2011
- 2013
- 2015
- 2016
- 2017
- 2018
- 2019
- 2020
- 2021
- 22
- 24
- 25
- 2D
- 30
- 300
- 35%
- 4
- 40
- 41
- 45
- 46
- 48
- 5
- 50
- 500
- 52
- 54
- 58
- 6
- 62
- 65
- 7
- 70
- 75
- 80
- 89
- 9
- a
- arriba
- Absoluto
- Conforme
- precisamente
- adquirir
- Actúe
- adaptado
- adicional
- la adición de
- adición
- unido
- Equilibrado
- administrado
- Después
- en contra
- agregado
- agregación
- AL
- Alexa
- Todos
- am
- cantidad
- an
- analizar
- análisis
- análisis
- Comercial
- y
- animal
- animales
- Antonio
- Anticuerpos
- anticuerpo
- antígeno
- aplicada
- aprobado
- agua
- somos
- artículo
- AS
- ficticio
- At
- automáticamente
- Hoy Disponibles
- b
- fondo
- BANDA
- Banca
- basado
- BD
- BE
- se convirtió en
- antes
- a continuación
- entre
- biomarcadores
- ciego
- bloqueo
- cuerpo
- ambas
- Fondo
- Cerebro
- Ruptura
- brevemente
- BSA
- buffer
- Construir la
- pero
- by
- calculado
- cámara
- Cáncer
- Células cancerígenas
- capaz
- Capacidad
- servicios sociales
- estudiar cuidadosamente
- llevado a
- (SCD por sus siglas en inglés),
- Células
- celulares
- el cambio
- cambiado
- caracterizada
- Charles
- elegido
- circular
- clase
- espacio
- clic
- cóctel
- código
- frío
- recogida
- --
- Financiamiento para la
- Columnas
- comparable
- en comparación con
- compliance
- cumpliendo
- concentración
- condición
- condiciones
- llevado a cabo
- conductible
- connolly
- Envase
- continuamente
- contribución
- control
- controles
- convencional
- cable
- Corona
- Consejo
- cubierta
- Cultura
- curva
- Corte
- Cycle
- Oscuro
- datos
- puntos de datos
- día
- Días
- de
- se define
- entrega
- densidad
- dependiente
- descrito
- Diseño
- detalle
- detectado
- Detección
- determinación
- Determinar
- determina
- determinar
- desarrollado
- el desarrollo
- Desarrollo
- HIZO
- un cambio
- una experiencia diferente
- digital
- diluido
- dilución
- descartado
- plato
- disperso
- distinto
- dividido
- DOE
- dosificación
- dosificar
- dibujo
- duplicados
- duración
- durante
- lugar de trabajo dinámico
- disfunción
- e
- E & T
- cada una
- Temprano en la
- efecto
- los efectos
- eficiencias
- eficiencia
- emisión
- facilita
- mejorado
- Mejora
- ecuación
- equipado
- Equivalente a
- se establece
- Éter (ETH)
- ético
- EV
- evaluar
- evaluado
- evaluación
- Cada
- evs
- experimento
- experimental
- experimentos
- expresados
- expresión
- hembra
- lleno
- filtrar
- filtración
- filtros
- final
- Nombre
- fijas
- de tus señales
- seguido
- siguiendo
- formaldehído
- Formalmente
- formación
- adelante
- Digital XNUMXk
- fracción
- FRAME
- Frecuencia
- fresco
- Desde
- función
- funcionalmente
- promover
- GAS
- cerrado
- gating
- generar
- suaves
- vaso
- grado
- gráficos
- Grupo procesos
- Grupo
- Cosecha
- Corazón
- Inicio
- Muebles para oficina
- HORAS
- viviendas
- HTTPS
- humana
- i
- HIELO
- Identificación
- identifica
- iii
- imagen
- Proyección de imagen
- inmune
- inmunidad
- inmunomodulador
- in
- incluido
- aumente
- incubado
- Incubando
- Incubación
- incubadora
- Individualmente
- induce
- inflamación
- inflamatorio
- información
- infusión
- Instrucciones
- instrumentos
- interacción
- intereses
- dentro
- intravenoso
- por vía intravenosa
- aislado
- solo
- mantenido
- riñón
- matanza
- kit
- el lab
- etiquetado
- Falta
- carente
- láser
- luego
- capas
- Nivel
- Licencia
- límites
- LINK
- vinculado
- para vivir
- Hígado
- alga viva
- Londres
- compromiso a largo plazo
- Baja
- luz
- macrófagos
- hecho
- mantener
- mantenido
- gran
- varón
- manual
- marcador
- Marte
- Misa
- materiales
- máximas
- personalizado
- mesurado
- multiplataforma
- medidas
- Medios
- servicios
- la investigación médica
- mediano
- mencionado
- metabólico
- Metanol
- Método
- métodos
- los métodos fueron
- ratones
- migración
- Leche
- min
- mínimo
- menor de edad
- mezcla
- mezclado
- mezcla
- ML
- movilidad
- Modificaciones
- MOL
- Morgan
- ratón
- Llamado
- nano
- nanotecnología
- Naturaleza
- no
- normal
- número
- números
- obtener
- obtenido
- obtención
- of
- Oficina
- Oficial
- Viejo
- on
- una vez
- ONE
- , solamente
- habiertos
- operado
- óptico
- óptimo
- or
- nuestros
- salir
- Más de
- durante la noche
- pasajes
- PBS
- para
- realizado
- Petri
- fenotipo
- piezas
- atravesar
- Platón
- Inteligencia de datos de Platón
- PlatónDatos
- más
- puntos
- polinomio
- población
- Portal
- portafolio
- La positividad
- Publicación
- preparación
- Preparar
- preparado
- anterior
- previamente
- primario
- Anterior
- procedimientos
- procedimientos
- proyecto
- proliferación
- promover
- proteger
- Cobertura
- Proteínas
- Proteínas
- protocolo
- probado
- publicado
- bomba
- cuantificación
- R
- Radiante
- proporción
- rbc
- alcanzado
- alcanzando
- reacción
- Leer
- Testimoniales
- recientemente
- receptor
- recomendaciones
- grabado
- reducir
- referencia
- reflejar
- regiones
- regulador
- relativo
- liberado
- eliminación
- remove
- Remoto
- renal
- reemplazados
- reemplazo
- Informes
- Requisitos
- la investigación
- respeto
- resultante
- Resultados
- revertir
- una estrategia SEO para aparecer en las búsquedas de Google.
- River
- Conferencia
- Ejecutar
- s
- sal
- mismo
- muestra
- SCI
- científico
- secundario
- seleccionado
- selección
- Sensibilidad
- Sepsis
- Serum
- set
- lado
- Signal
- señales
- importante
- Silver
- Del mismo modo
- soltero
- Tamaño
- tamaños
- desnatar
- Piel
- Despacio
- chica
- So
- sodio
- Software
- a medida
- Soluciones
- velocidad
- Girar
- giros
- estándar
- estadístico
- estadísticamente
- Stealth
- Potencias
- células madre
- paso
- Detener
- detenido
- STORAGE
- almacenados
- estudios
- ESTUDIO
- sustrato
- Superficie
- suspensión
- te
- T
- toma
- toma
- orientación
- test
- probado
- esa
- El proyecto
- el Reino Unido
- su
- luego
- Ahí.
- así
- Tres
- tridimensional
- umbral
- A través de esta formación, el personal docente y administrativo de escuelas y universidades estará preparado para manejar los recursos disponibles que derivan de la diversidad cultural de sus estudiantes. Además, un mejor y mayor entendimiento sobre estas diferencias y similitudes culturales permitirá alcanzar los objetivos de inclusión previstos.
- veces
- a
- Total
- Trazador
- Formación
- transferido
- tratados
- Twice
- dos
- tipo
- tipos
- Uk
- bajo
- unidad
- hasta
- a
- consumo
- usado
- usando
- Valores
- veterinario
- vía
- viabilidad
- Video
- vivo
- volumen
- fue
- Lavado
- Agua
- Watson
- longitudes de onda
- semana
- Semanas
- Bienestar
- WELL
- tuvieron
- West
- rodador
- cuando
- que
- complejo de salvador blanco
- dentro de
- sin
- los rendimientos
- zephyrnet
- zombi