180,000D 構造が決定されたユニークなタンパク質は 3 を超えています。 毎年何万もの新しい構造物が解決されています。これはほんの一部にすぎません 特徴的な配列を持つ 200 億個の既知のタンパク質。最近の深層学習アルゴリズムには、 アルファフォールド は、タンパク質の配列を使用してタンパク質の 3D 構造を正確に予測できるため、タンパク質の 3D 構造データを数百万単位に拡張するのに役立ちます。グラフ ニューラル ネットワーク (GNN) は、アミノ酸残基のグラフで表現できるタンパク質構造から情報を抽出する効果的な深層学習アプローチとして登場しました。個々のタンパク質グラフには通常、数百のノードが含まれており、管理可能なサイズです。何万ものタンパク質グラフを、次のようなシリアル化されたデータ構造に簡単に保存できます。 TFレコード GNN のトレーニング用。ただし、何百万ものタンパク質構造に対して GNN をトレーニングするのは困難です。データのシリアル化は、テラバイト規模のデータセット全体をメモリに読み込む必要があるため、数百万のタンパク質構造に拡張できません。
この投稿では、に保存されている何百万ものタンパク質で GNN をトレーニングできる、スケーラブルな深層学習ソリューションを紹介します。 Amazon DocumentDB(MongoDB互換性あり) アマゾンセージメーカー.
説明の目的で、実験的に決定された公的に入手可能なタンパク質構造を使用します。 タンパク質データバンク そして、そこからコンピューターによってタンパク質構造を予測します。 AlphaFoldタンパク質構造データベース。機械学習 (ML) の問題は、3D 構造から構築されたタンパク質グラフに基づいて実験構造と予測構造を区別するための識別子 GNN モデルを開発することです。
ソリューションの概要
まずタンパク質の構造を解析して、n 次元配列やネストされたオブジェクトなどの複数のタイプのデータ構造を含む JSON レコードを作成し、タンパク質の原子座標、プロパティ、識別子を保存します。タンパク質の構造の JSON レコードの保存には平均 45 KB かかります。 100 億個のタンパク質を保存するには約 4.2 TB が必要になると予測しています。 Amazon DocumentDB ストレージ データに合わせて自動的にスケールします クラスター ボリュームに 10 GB 単位で最大 64 TB まで追加できます。したがって、JSON データ構造とスケーラビリティのサポートにより、Amazon DocumentDB が自然な選択となります。
次に、構造から構築されたアミノ酸残基のグラフを使用してタンパク質の特性を予測するための GNN モデルを構築します。 GNN モデルは SageMaker を使用してトレーニングされ、データベースからタンパク質構造のバッチを効率的に取得するように構成されています。
最後に、トレーニングされた GNN モデルを分析して、予測についての洞察を得ることができます。
このチュートリアルでは次の手順を実行します。
- を使用してリソースを作成します AWS CloudFormation テンプレート。
- タンパク質の構造とプロパティを準備し、データを Amazon DocumentDB に取り込みます。
- SageMaker を使用してタンパク質構造について GNN をトレーニングします。
- トレーニングされた GNN モデルをロードして評価します。
この投稿で使用されているコードとノートブックは、次の場所から入手できます。 GitHubレポ.
前提条件
このチュートリアルでは、次の前提条件を満たしている必要があります。
このチュートリアルを 2.00 時間実行しても、費用は XNUMX ドル以下になります。
リソースを作成する
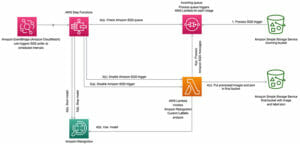
我々は CloudFormationテンプレート この投稿に必要な AWS リソースを、投稿と同様のアーキテクチャで作成します。 Amazon SageMaker を使用した Amazon DocumentDB (MongoDB 互換) に保存されたデータの分析。 CloudFormation スタックの作成手順については、ビデオを参照してください。 AWS CloudFormation を使用してインフラストラクチャ管理を簡素化する.
CloudFormation スタックは以下をプロビジョニングします。
- Amazon DocumentDB 用の 3 つのプライベート サブネットと、それぞれ SageMaker ノートブック インスタンスと ML トレーニング コンテナ用の 2 つのパブリック サブネットを持つ VPC。
- 各プライベートサブネットに 1 つずつ、合計 3 つのノードを持つ Amazon DocumentDB クラスター。
- Amazon DocumentDB のログイン認証情報を保存するための Secrets Manager シークレット。これにより、平文の認証情報を SageMaker インスタンスに保存することを回避できます。
- データを準備し、トレーニング ジョブを調整し、対話型分析を実行するための SageMaker ノートブック インスタンス。
CloudFormation スタックを作成するときは、以下を指定する必要があります。
- CloudFormation スタックの名前
- Amazon DocumentDB のユーザー名とパスワード (Secrets Manager に保存される)
- Amazon DocumentDB インスタンスタイプ (デフォルトは db.r5.large)
- SageMaker インスタンス タイプ (デフォルト ml.t3.xlarge)
CloudFormation スタックの作成には約 15 分かかります。次の図は、リソース アーキテクチャを示しています。
タンパク質の構造とプロパティを準備し、データを Amazon DocumentDB に取り込みます
このセクションの後続のコードはすべて Jupyter ノートブックにあります Prepare_data.ipynb CloudFormation スタックで作成された SageMaker インスタンス内。
このノートブックは、タンパク質構造データを準備して Amazon DocumentDB に取り込むために必要な手順を処理します。
- まず、予測されたタンパク質構造を以下からダウンロードします。 アルファフォールド DB PDB 形式と、一致する実験構造 タンパク質データバンク.
デモンストレーションの目的で、好熱性古細菌のタンパク質のみを使用します。 メタノカルドコッカス・ジャンナスキーこれは、私たちが扱うことができる 1,773 個のタンパク質からなる最小のプロテオームを持っています。他の種のタンパク質を試してみるのも大歓迎です。
- Secrets Manager に保存されている認証情報を取得して、Amazon DocumentDB クラスターに接続します。
- Amazon DocumentDB への接続を設定した後、PDB ファイルを JSON レコードに解析してデータベースに取り込みます。
PDB ファイルの解析に必要なユーティリティ関数を提供します。 pdb_parse.pyを選択します。 parse_pdb_file_to_json_record この関数は、PDB ファイル内の 1 つまたは複数のペプチド鎖から原子座標を抽出するという重労働を実行し、1 つまたは JSON ドキュメントのリストを返します。これは、ドキュメントとして Amazon DocumentDB コレクションに直接取り込むことができます。次のコードを参照してください。
解析されたタンパク質データを Amazon DocumentDB に取り込んだ後、タンパク質ドキュメントの内容を更新できます。たとえば、タンパク質構造をトレーニング、検証、またはテスト セットで使用する必要があるかどうかを示すフィールドを追加すると、モデルのトレーニング ロジスティクスが簡単になります。
- まず、フィールドを持つすべてのドキュメントを取得します。
is_AF集約パイプラインを使用してドキュメントを階層化するには:
- 次に、
update_many分割情報を Amazon DocumentDB に保存する関数:
SageMaker を使用してタンパク質構造について GNN をトレーニングする
このセクションの後続のコードはすべて、 Train_and_eval.ipynb CloudFormation スタックで作成された SageMaker インスタンスのノートブック。
このノートブックは、Amazon DocumentDB に保存されているタンパク質構造データセットで GNN モデルをトレーニングします。
まず、Amazon DocumentDB からタンパク質ドキュメントのミニバッチを取得できるタンパク質データセット用の PyTorch データセット クラスを実装する必要があります。組み込みのプライマリ ID (_id).
- を拡張して反復可能なスタイルのデータセットを使用します。 IterableDatasetをプリフェッチします。
_id初期化時のドキュメントのラベル:
-
ProteinDatasetでデータベース読み取り操作を実行します。__iter__方法。複数のワーカーがある場合、ワークロードを均等に分割しようとします。
- 上記
__iter__このメソッドは、タンパク質の原子座標も次のように変換します。 DGLグラフ Amazon DocumentDB からロードされた後のオブジェクトconvert_to_graph関数。この関数は、C アルファ原子の 3D 座標を使用してアミノ酸残基の k 近傍 (kNN) グラフを構築し、残基の同一性を表すワンホット エンコードされたノード特徴を追加します。
-
ProteinDataset実装すると、データセットのトレーニング、検証、テスト用にインスタンスを初期化し、トレーニング インスタンスをラップすることができます。BufferedShuffleDatasetシャッフルを有効にします。 - さらにそれらを包みます
torch.utils.data.DataLoaderの他のコンポーネントを操作するには SageMaker PyTorch エスティメーター トレーニングスクリプト。 - 次に、解釈を容易にするために、グローバル アテンション プーリング層を備えた単純な 2 層のグラフ畳み込みネットワーク (GCN) を実装します。
- その後、この GCN をトレーニングできます。
ProteinDatasetタンパク質構造が AlphaFold によって予測されるかどうかを予測するバイナリ分類タスクのインスタンスです。目的関数としてバイナリ クロス エントロピーを使用し、確率的勾配最適化には Adam オプティマイザーを使用します。完全なトレーニング スクリプトは次の場所にあります。 src/main.py.
次に、トレーニング ジョブを処理するために SageMaker PyTorch Estimator をセットアップします。 SageMaker によって開始されたマネージド Docker コンテナが Amazon DocumentDB に接続できるようにするには、Estimator のサブネットとセキュリティ グループを設定する必要があります。
- サブネット ID を取得します。 ネットワーク アドレス変換 (NAT) ゲートウェイ 存在するものと、Amazon DocumentDB クラスターの名前別のセキュリティ グループ ID です。
トレーニングされた GNN モデルをロードして評価する
トレーニング ジョブが完了したら、トレーニングされた GCN モデルをロードし、詳細な評価を実行できます。
次の手順のコードはノートブックにもあります。 Train_and_eval.ipynb.
SageMaker トレーニング ジョブは、モデル アーティファクトをデフォルトの S3 バケットに保存します。このバケットの URI には、 estimator.model_data 属性。に移動することもできます。 トレーニングの仕事 SageMaker コンソールのページにアクセスして、評価するトレーニング済みモデルを見つけます。
- 研究目的で、モデル アーティファクト (学習したパラメータ) を PyTorch にロードできます。 state_dict 次の関数を使用します。
- 次に、精度を計算することにより、完全なテスト セットに対して定量的モデル評価を実行します。
GCN モデルは 74.3% の精度を達成しましたが、クラス事前確率に基づいて予測を行うダミーのベースライン モデルは 56.3% しか達成できなかったことがわかりました。
また、GCN モデルの解釈可能性にも関心があります。グローバル アテンション プーリング レイヤーを実装しているため、ノード全体のアテンション スコアを計算して、モデルによって行われた特定の予測を説明できます。
- 次に、注意スコアを計算し、同じペプチドからの構造のペア (AlphaFold 予測と実験) のタンパク質グラフに重ね合わせます。
上記のコードは、ノード上の注意スコアを重ねた次のタンパク質グラフを生成します。モデルのグローバル注意プーリング層は、タンパク質構造が AlphaFold によって予測されるかどうかを予測するために重要であるとして、タンパク質グラフ内の特定の残基を強調表示できることがわかりました。これは、これらの残基が予測タンパク質構造と実験タンパク質構造において特徴的なグラフ トポロジーを持っている可能性があることを示しています。
 |
 |
要約すると、Amazon DocumentDB に保存されているタンパク質構造で GNN をトレーニングするためのスケーラブルな深層学習ソリューションを紹介します。このチュートリアルではトレーニングに数千のタンパク質のみを使用しますが、このソリューションは数百万のタンパク質に拡張可能です。タンパク質データセット全体をシリアル化するなどの他のアプローチとは異なり、私たちのアプローチはメモリを大量に使用するワークロードをデータベースに転送するため、トレーニング ジョブのメモリが複雑になります。 O(batch_size)、トレーニングするタンパク質の総数には依存しません。
クリーンアップ
今後の料金発生を避けるために、作成した CloudFormation スタックを削除してください。これにより、VPC、Amazon DocumentDB クラスター、SageMaker インスタンスなど、CloudFormation テンプレートを使用してプロビジョニングしたすべてのリソースが削除されます。手順については、を参照してください。 AWSCloudFormationコンソールでスタックを削除する.
まとめ
私たちは、タンパク質構造を Amazon DocumentDB に保存し、SageMaker からデータのミニバッチを効率的に取得することで、数百万のタンパク質構造に拡張可能なクラウドベースの深層学習アーキテクチャについて説明しました。
タンパク質の特性予測における GNN の使用について詳しくは、最近の出版物をご覧ください。 LM-GVP、配列と構造からタンパク質の特性を予測するための一般化可能な深層学習フレームワーク.
著者について
 王子辰博士号は、Amazon Machine Learning Solutions Lab の応用科学者です。生物学的データや医療データを使用した ML および統計手法の開発における数年間の研究経験を活かし、さまざまな業界の顧客と協力して ML の問題を解決しています。
王子辰博士号は、Amazon Machine Learning Solutions Lab の応用科学者です。生物学的データや医療データを使用した ML および統計手法の開発における数年間の研究経験を活かし、さまざまな業界の顧客と協力して ML の問題を解決しています。
 セルバン・センティベル は、AWSのAmazon ML Solutions LabのシニアMLエンジニアであり、機械学習、ディープラーニングの問題、エンドツーエンドのMLソリューションについてお客様を支援することに重点を置いています。 彼はAmazonComprehendMedicalの創設エンジニアリングリーダーであり、複数のAWSAIサービスの設計とアーキテクチャに貢献しました。
セルバン・センティベル は、AWSのAmazon ML Solutions LabのシニアMLエンジニアであり、機械学習、ディープラーニングの問題、エンドツーエンドのMLソリューションについてお客様を支援することに重点を置いています。 彼はAmazonComprehendMedicalの創設エンジニアリングリーダーであり、複数のAWSAIサービスの設計とアーキテクチャに貢献しました。
- '
- "
- 000
- 100
- 11
- 3d
- 7
- 9
- AI
- AIサービス
- アルゴリズム
- すべて
- Amazon
- Amazon Comprehend
- アマゾン機械学習
- アマゾンセージメーカー
- 建築
- 周りに
- AWS
- ベースライン
- ボディ
- ビルド
- 課金
- 分類
- コード
- 計算
- 接続
- 領事
- コンテナ
- コンテナ
- 中身
- 貢献
- 作成
- Credentials
- Customers
- データ
- データベース
- 深い学習
- 設計
- 開発する
- デッカー
- Dockerコンテナ
- ドキュメント
- 効果的な
- エンジニア
- エンジニアリング
- 体験
- 特徴
- 特徴
- イチジク
- 最後に
- 名
- 形式でアーカイブしたプロジェクトを保存します.
- フォワード
- フレームワーク
- フル
- function
- 未来
- ギャップ
- グローバル
- グループ
- 特徴
- HTTPS
- 含めて
- 情報
- インフラ
- 洞察
- 相互作用的
- IT
- ジョブ
- Jobs > Create New Job
- ジュピターノート
- キー
- ラベル
- 大
- つながる
- LEARN
- 学んだ
- 学習
- リスト
- 負荷
- 物流
- 機械学習
- 作成
- 管理
- ゲレンデマップ
- math
- 医療の
- 百万
- ML
- MongoDBの
- net
- ネットワーク
- ニューラル
- ニューラルネットワーク
- ノード
- ノートPC
- その他
- パスワード
- 平文
- PoS
- 予測
- 予測
- プライベート
- 生産された
- プロジェクト
- 財産
- タンパク質
- 公共
- パイトーチ
- 記録
- 研究
- リソースを追加する。
- リソース
- 収益
- ラン
- セージメーカー
- スケーラビリティ
- 規模
- セキュリティ
- サービス
- セッションに
- 簡単な拡張で
- サイズ
- 小さい
- ソリューション
- 解決する
- split
- start
- 都道府県
- ストレージ利用料
- 店舗
- サポート
- テンソルフロー
- test
- トーチ
- トレーニング
- 列車
- インタビュー
- チュートリアル
- アップデイト
- URI
- us
- ユーティリティ
- ビデオ
- 可視化
- ボリューム
- Wikipedia
- 以内
- 仕事
- 労働者
- 作品
- 年
- ユーチューブ