Introductie
Afhankelijk van de sector en het specifieke voorbeeld, omvat anomaliedetectie het opsporen van ongebruikelijke of onregelmatige patronen in gegevens om ongewenste of vreemde gebeurtenissen op te sporen.
Anomaliedetectie kan helpen bij het zien van pieken in gedeeltelijk voltooide of volledig voltooide transacties in sectoren zoals e-commerce, marketing en andere, waardoor het mogelijk wordt om in te spelen op verschuivingen in de vraag of om te dure dingen te ontdekken.
Detectie van afwijkingen is belangrijk in de gezondheidszorg om potentiële gezondheidsrisico's op te sporen, letsel te voorkomen en de patiëntenzorg te verbeteren. Het kan helpen bij het bewaken van de statistieken van chronische aandoeningen van patiënten, het identificeren van frauduleuze medische claims en het gebruik van medische beeldvorming om ziekten te diagnosticeren.
Kortom, afwijkingsdetectie is een cruciaal hulpmiddel in de gezondheidszorg dat helpt bij het identificeren van potentiële gezondheidsproblemen en het verbeteren van de patiëntenzorg.
Leerdoel
Dit project beoogt een grondig inzicht te verschaffen in ECG-signalen, hun gebruik bij anomaliedetectie en hun gebruik in de gezondheidszorg. Deelnemers leren over het filteren van tijdreeksgegevens en het belang van het kiezen van de beste methode voor hun gebruiksscenario. Bovendien zullen ze praktische ervaring hebben met het extraheren van kenmerken uit ECG-gegevens en ontdekken hoe deze kenmerken de effectiviteit van het model beïnvloeden. De constructie, training en evaluatie van een deep learning-model voor anomaliedetectie, zoals een LSTM, komen allemaal aan bod in dit project. Het belang van feature engineering en vakkennis zal worden benadrukt als essentiële elementen bij het creëren van efficiënte modellen voor tijdreeksgegevens. De lezer leert algoritmen voor machine learning te gebruiken om ECG-waarden te analyseren en onregelmatigheden in de praktijk op te sporen.
.
Dit artikel is gepubliceerd als onderdeel van het Data Science-blogathon.
Inhoudsopgave
Projectpijplijn
Het project omvat verschillende stappen, die hieronder worden beschreven. De code voor elke stap wordt in de volgende sectie onderzocht.
- Gegevensextractie: bij deze stap worden de ECG-gegevens geëxtraheerd TensorFlow datasets en laden deze in de werkruimte.
- Verkennende data-analyse: deze stap omvat het analyseren en visualiseren van de dataset om ontbrekende waarden te controleren. We zullen de normale en afwijkende ECG-signalen visualiseren om de trends te identificeren.
- Voorverwerking van gegevens: in deze stap worden de gegevens voorbereid voor modeltraining en validatie. De gegevens worden opgesplitst in kenmerken en labels, en de ECG-signalen worden gefilterd met behulp van verschillende algoritmen om de methode te kiezen op basis van Mean Squared Error-meting.
- Opsplitsen van trein en testset: we splitsen de gegevens op in een trainingsset en een testset voor modelevaluatiedoeleinden.
- Functie-extractie: dit is een cruciale stap in het project. We halen relevante kenmerken uit de tijdreeksgegevens om het model te trainen, aangezien zinvolle kenmerkextractie een grote invloed heeft op de prestaties van het model.
- Deep learning-model bouwen en trainen: we zullen de architectuur van het LSTM deep learning-model bouwen en de nodige parameters instellen, gevolgd door het trainen van het model.
- Modeltesten: Het model wordt getest door zijn voorspellingen te berekenen op basis van de testgegevens.
- Modelevaluatie en visualisaties: Het model wordt beoordeeld op basis van zijn voorspellingen op basis van de testgegevens en de resultaten worden geanalyseerd. Bovendien wordt de trainings- en valideringsfout gevisualiseerd na elke periode van modeltraining.
Voorwaarden
De voorwaarden om dit project te voltooien zijn:
- Vaardigheid in python.
- Basisprincipes van numpy, panda's, sklearn, scipy, Keras en Plotly.
- Basisprincipes van diep leren, met name het LSTM-model.
Gegevenssetbeschrijving
De ECG-dataset heeft 4998 voorbeelden met 140 tijdpunten en 1 doelvariabele.
Elke rij vertegenwoordigt een ECG-signaal en de waarden in de kolommen zijn de spanningsniveaus op elk tijdstip.
De laatste kolom is de doelvariabele met twee waarden: 1 voor normaal ECG en 0 voor afwijkend ECG.
Alle kolommen hebben zwevende gegevenstypen en de gegevensset heeft geen ontbrekende waarden.
Nu we inzicht hebben in de probleemstelling en de gegevens. Laten we doorgaan en beginnen met coderen.
Projectcode
De projectcode is gestructureerd in een stapsgewijze indeling, waardoor deze gemakkelijk te volgen is. Elke stap is onderverdeeld in subsecties om het begrip van de code te vereenvoudigen.
Stap 1: Importeer alle benodigde bibliotheken
Allereerst gaan we de bibliotheken importeren
#Importeer alle bibliotheken importeer panda's als pd importeer numpy als np van sklearn.preprocessing importeer MinMaxScaler importeer plotly.graph_objs as go importeer plotly.express als px van scipy.signal importeer medfilt, butter, filtfilt importeer pywt van sklearn.model_selection import train_test_split import scipy.signal van keras.models import Sequential van keras.layers import LSTM, Dense, Reshape van sklearn.metrics import
Stap 2: gegevensextractie
Deze stap omvat het verkrijgen van de dataset en het laden ervan in uw python-omgeving.
#importeer de dataset van tensorflow datasets df = pd.read_csv('http://storage.googleapis.com/download.tensorflow.org/data/ecg.csv', header=None)
Stap 3: Verkennende gegevensanalyse
Laten we nu de gegevens onderzoeken door de vorm, gegevenstypen en de aanwezigheid van ontbrekende waarden te controleren. Daarnaast zullen we de afwijkende en normale ECG-signalen visualiseren om te zoeken naar opvallende patronen.
1. Geef de eerste 5 rijen met gegevens weer
df.head ()
Output:
2. De vorm van de dataset
df.vorm
Output:
(4998, 141)
3. Dataframe-informatie
df.info()
Output:
RangeIndex: 4998 ingangen, 0 tot 4997 Kolommen: 141 ingangen, 0 tot 140 dtypes: float64(141) geheugengebruik: 5.4 MB
4. Geef alle kolomnamen weer
df.kolommen
Output:
Int64Index([ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, ... 131, 132, 133, 134, 135, 136, 137, 138, 139, 140], dtype=' int64', lengte=141)
5. Labels van het type ECG-signalen
Geef de unieke waarden van de doelvariabele weer, de laatste kolom met een index "140".
df[140].uniek()
output:
matrix([1., 0.])
Geef het aantal voorbeelden voor elk label weer: afwijkend en normaal ECG.
df[140].value_counts() # 1 is normaal & 0 is abnormaal ecg
Output:
1.0 2919 0.0 2079 Naam: 140, dtype: int64
6. Geef de gegevenstypen voor elke kolom weer
df.dtypes
Output:
0 float64 1 float64 2 float64 3 float64 4 float64 ... 136 float64 137 float64 138 float64 139 float64 140 float64 Lengte: 141, dtype: object
7. Controleer of de gegevens ontbrekende waarden bevatten
Aantal ontbrekende waarden per kolom
#kijken naar ontbrekende waarden voor elke kolom df.isna().sum()
Output:
0 0 1 0 2 0 3 0 4 0 .. 136 0 137 0 138 0 139 0 140 0 Lengte: 141, dtype: int64
Ontbrekende waarden voor de gehele dataset
#ontbrekende waarden voor hele dataset df.isna().sum().sum()
Output:
0
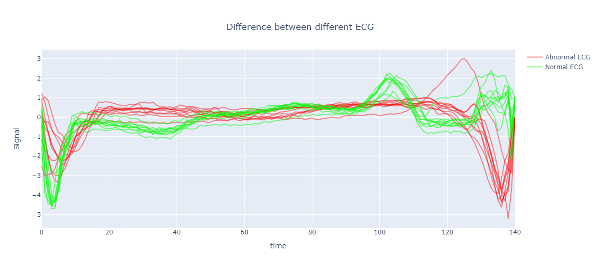
8. Normale en afwijkende ECG-signalen uitzetten
Laten we zowel de afwijkende als de normale ECG-signalen visualiseren. We zullen de dataset verdelen in normaal en afwijkend ECG en vervolgens 10 voorbeelden plotten met behulp van de Plotly-bibliotheek.
#plot grafieken van normaal en abnormaal ECG om de trends te visualiseren abnormaal = df[df.loc[:,140] ==0][:10] normaal = df[df.loc[:,140] ==1][: 10] # Maak de figuur fig = go.Figuur() #maak een lijst om slechts één legenda weer te geven leg = [False] * abnormal.shape[0] leg[0] = True
# Plottraining en validatiefout voor i in bereik(abnormal.shape[0]): fig.add_trace(go.Scatter( x=np.arange(abnormal.shape[1]),y=abnormal.iloc[i,: ],name='Abnormaal ECG', mode='lijnen', marker_color='rgba(255, 0, 0, 0.9)', showlegend= leg[i])) voor j in range(normal.shape[0]) : fig.add_trace(go.Scatter( x=np.arange(normal.shape[1]),y=normal.iloc[j,:],name='Normal ECG', mode='lines', marker_color=' rgba(0, 255, 0, 1)', showlegend= leg[j])) fig.update_layout(xaxis_title="time", yaxis_title="Signal", title= {'text': 'Verschil tussen verschillende ECG', 'xanchor': 'center', 'yanchor': 'top', 'x':0.5} , bargap=0,) fig.update_traces(dekking=0.5) fig.show()
Uitvoergrafiek:
Stap 4: Voorverwerking van gegevens
Om de gegevens voor ons machine-learningmodel voor te bereiden, moeten we deze voorbewerken. Dit omvat het splitsen van de gegevens in labels en functies, het normaliseren van de dataset en het verwijderen van ruis uit het ECG-signaal met behulp van verschillende filteralgoritmen. We zullen de resultaten evalueren met behulp van visualisatie en foutberekening om het beste filteralgoritme te bepalen.
1. Splits de dataset op in features en labels
# splits de data op in labels en features ecg_data = df.iloc[:,:-1] labels = df.iloc[:,-1]
2. Normaliseer de dataset
# Normaliseer de gegevens tussen -1 en 1 scaler = MinMaxScaler(feature_range=(-1, 1)) ecg_data = scaler.fit_transform(ecg_data)
3. Filter de dataset
Laten we nu de gegevens filteren om ruis te elimineren. We zullen drie algoritmen gebruiken: mediaanfiltering, laagdoorlaatfiltering en waveletfiltering.
#filtering van de onbewerkte signalen # Mediaan filtering ecg_medfilt = medfilt(ecg_data, kernel_size=3)
# Low-pass filtering lowcut = 0.05 highcut = 20.0 nyquist = 0.5 * 360.0 low = lowcut / nyquist high = highcut / nyquist b, a = butter(4, [low, high], btype='band') ecg_lowpass = filtfilt( b, a, ecg_data)
# Wavelet filtering coeffs = pywt.wavedec(ecg_data, 'db4', level=1) drempelwaarde = np.std(coeffs[-1]) * np.sqrt(2*np.log(len(ecg_data))) coeffs[ 1:] = (pywt.threshold(i, value=threshold, mode='soft') voor i in coeffs[1:]) ecg_wavelet = pywt.waverec(coeffs, 'db4')
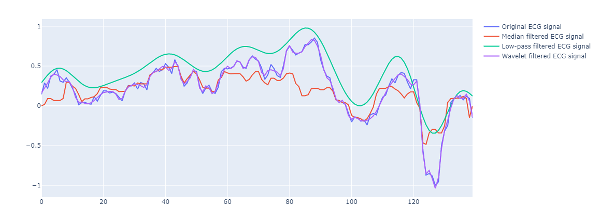
4. Teken de grafieken van ongefilterde en gefilterde signalen
Laten we, voor visuele vergelijking, de ongefilterde en gefilterde signalen in een enkele grafiek uitzetten. Deze visualisatie helpt bij het selecteren van het meest effectieve filteralgoritme door naar de grafiek te kijken.
# Plot origineel ECG-signaal fig = go.Figuur() fig.add_trace(go.Scatter(x=np.arange(ecg_data.shape[0]), y=ecg_data[30], mode='lines', name=' Origineel ECG-signaal')) # Plot gefilterde ECG-signalen fig.add_trace(go.Scatter(x=np.arange(ecg_medfilt.shape[0]), y=ecg_medfilt[30], mode='lines', name='Median gefilterd ECG-signaal')) fig.add_trace(go.Scatter(x=np.arange(ecg_lowpass.shape[0]), y=ecg_lowpass[30], mode='lines', name='Low-pass gefilterd ECG-signaal ')) fig.add_trace(go.Scatter(x=np.arange(ecg_wavelet.shape[0]), y=ecg_wavelet[30], mode='lines', name='Wavelet gefilterd ECG-signaal')) fig. show()
Uitvoergrafiek:
5. De beste filtertechniek kiezen
We zullen de filtermethoden evalueren door de gemiddelde kwadratische fout (MSE) van elk algoritme te berekenen. Het algoritme met de laagste MSE wordt geselecteerd.
Om MSE te berekenen, moet het dataframe van beide signalen een gelijk aantal rijen hebben.
Als de gegevensset na het filteren wordt verkleind, omvat deze stap het opvullen van de gegevens met nullen.
#pad het signaal met nullen
def pad_data(original_data,filtered_data): # Bereken het verschil in lengte tussen de originele data en gefilterde data diff = original_data.shape[1] - filtered_data.shape[1] # vul de kortere array op met nullen als diff > 0: # Create een reeks nullen met dezelfde vorm als de oorspronkelijke gegevensopvulling = np.zeros((filtered_data.shape[0], originele_data.shape[1])) # Voeg de gefilterde gegevens samen met de opvulling opgevulde_data = np.concatenate((filtered_data , opvulling)) elif diff < 0: opgevulde_data = gefilterde_data[:,:-abs(diff)] elif diff == 0: opgevulde_data = gefilterde_data return opgevulde_data
Vervolgens berekenen we de gemiddelde kwadratische fout voor alle technieken.
def mse(originele_gegevens, gefilterde_gegevens):
filter_data = pad_data(original_data,filtered_data) return np.mean((original_data - filter_data) ** 2) # Bereken MSE mse_value_m = mse(ecg_data, ecg_medfilt) mse_value_l = mse(ecg_data, ecg_lowpass) mse_value_w = mse(ecg_data, ecg_wavelet) print ("MSE-waarde van mediaanfiltering:", mse_value_m) print("MSE-waarde van laagdoorlaatfiltering:", mse_value_l) print("MSE-waarde van waveletfiltering:", mse_value_w)
Output:
MSE-waarde van mediaanfiltering: 0.017260298402611125 MSE-waarde van laagdoorlaatfiltering: 0.36750805414756493 MSE-waarde van waveletfiltering: 0.0010818752598698714
Op basis van de hierboven weergegeven MSE-waarde wordt wavelet-filtering gekozen.
Stap 5: gegevens splitsen in trein- en testset
De dataset is verdeeld in 80% voor training en 20% voor test- en validatiedoeleinden.
# De gegevens splitsen in trein- en testsets X_train, X_test, y_train, y_test = train_test_split(ecg_wavelet, labels, test_size=0.2, random_state=42)
Stap 6: Functie-extractie
Wat zijn de kenmerken?
Kenmerken zijn de attributen die bepaalde gegevens beschrijven en kunnen worden gebruikt om voorspellingen te doen of ongebruikelijke gevallen te classificeren. Het is belangrijk om relevante kenmerken te extraheren, omdat het invoeren van irrelevante of overbodige informatie aan het model een negatieve invloed kan hebben op de prestaties en nauwkeurigheid ervan.
Waarom functies extraheren?
Na het voorbewerken van de gegevens, moeten we kenmerken uit de ECG-signalen halen voor ons deep learning-model. Het rechtstreeks trainen van het model op alle tijdreeksgegevens kan leiden tot overfitting en kan rekenkundig duur zijn. We kunnen de hoeveelheid gegevens verminderen door relevante kenmerken te extraheren en het generalisatievermogen van het model naar nieuwe, ongeziene gegevens te verbeteren.
Relevante functies kiezen
In deze context worden de volgende functies gebruikt:
- T-amplitude: de hoogte van de T-golf op de grafiek van het elektrocardiogram (ECG), die de terugkeer van het hart naar rust na een samentrekking symboliseert
- R-amplitude: de hoogte van de R-golf op de ECG-grafiek vertegenwoordigt de initiële samentrekking van de hartspier.
- RR-interval: de tijd tussen twee opeenvolgende R-golven op de ECG-grafiek, die de tijd tussen hartslagen weergeeft.
- QRS-duur: de tijd die nodig is voor het QRS-complex, dat het elektrische signaal vertegenwoordigt dat door de ventrikels gaat.
Proces van kenmerkextractie:
Om deze kenmerken te extraheren, berekenen we eerst de R & T-pieken, de R-amplitude, het RR-interval, enz. Met behulp van de scipy.signals-bibliotheek. Vervolgens berekenen we het gemiddelde, de mediaan, de som en andere statistische statistieken van elk kenmerk om de kenmerken vast te leggen. Al deze functies worden vervolgens opgeslagen in een array.
5. Kenmerkextractie van het treinstel
# Een lege lijst initialiseren om de kenmerken op te slaan kenmerken = [] # Kenmerken extraheren voor elk monster voor i in bereik (X_train.shape [0]): # De R-pieken vinden r_peaks = scipy.signal.find_peaks (X_train [i] )[0] #Initialiseer lijsten om R-piek- en T-piekamplitudes vast te houden r_amplitudes = [] t_amplitudes = [] # Doorloop R-pieklocaties om overeenkomstige T-piekamplitudes te vinden voor r_peak in r_peaks: # Zoek de index van de T-piek (minimumwaarde) in het interval van R-piek tot R-piek + 200 monsters t_peak = np.argmin(X_train[i][r_peak:r_peak+200]) + r_peak #Voeg de R-piekamplitude en T toe -piekamplitude naar de lijsten r_amplitudes.append(X_train[i][r_peak]) t_amplitudes.append(X_train[i][t_peak]) # het extraheren van singuliere waarden uit de r_amplitudes std_r_amp = np.std(r_amplitudes) mean_r_amp = np. mean(r_amplitudes) median_r_amp = np.median(r_amplitudes) sum_r_amp = np.sum(r_amplitudes) # het extraheren van singuliere waarden uit de t_amplitudes std_t_amp = np.std(t_amplitudes) mean_t_amp = n p.mean(t_amplitudes) median_t_amp = np.median(t_amplitudes) sum_t_amp = np.sum(t_amplitudes) # Zoek de tijd tussen opeenvolgende R-pieken rr_intervals = np.diff(r_peaks) # Bereken de tijdsduur van de gegevensverzameling time_duration = (len(X_train[i]) - 1) / 1000 # ervan uitgaande dat gegevens in ms zijn # Bereken de bemonsteringssnelheid bemonsteringssnelheid = len(X_trein[i]) / tijd_duur # Bereken hartslagduur = len(X_trein[i]) / bemonsteringssnelheid heart_rate = (len(r_peaks) / duur) * 60 # QRS-duur qrs_duration = [] voor j in bereik(len(r_peaks)): qrs_duration.append(r_peaks[j]-r_peaks[j-1]) # extraheren singuliere waarde metrics from the qrs_durations std_qrs = np.std(qrs_duration) mean_qrs = np.mean(qrs_duration) median_qrs = np.median(qrs_duration) sum_qrs = np.sum(qrs_duration) # De singuliere waarden metrics extraheren uit het RR-interval std_rr = np .std(rr_intervals) mean_rr = np.mean(rr_intervals) median_rr = np.median(rr_intervals) sum_rr = np.sum(rr_intervals) # De algemene standaard extraheren d afwijking std = np.std(X_train[i]) # Extraheren van het algehele gemiddelde gemiddelde = np.mean(X_train[i]) # Toevoegen van de kenmerken aan de lijst features.append([mean, std, std_qrs, mean_qrs,median_qrs , sum_qrs, std_r_amp, mean_r_amp, median_r_amp, sum_r_amp, std_t_amp, mean_t_amp, median_t_amp, sum_t_amp, sum_rr, std_rr, mean_rr,median_rr, heart_rate]) # De lijst converteren naar een numpy array features = np.array(features)
We hebben nu 19 kenmerken uit de dataset gehaald.
De vorm van deze trainingsset na kenmerkextractie is:
(3998, 19)
6. Functie-extractie van de testset
Op dezelfde manier zullen we de kenmerken van de testset extraheren.
# Een lege lijst initialiseren om de kenmerken op te slaan X_test_fe = [] # Kenmerken extraheren voor elk monster voor i in bereik (X_test.shape [0]): # De R-pieken vinden r_peaks = scipy.signal.find_peaks (X_test [i] )[0] # Lijsten initialiseren om R-piek- en T-piekamplitudes vast te houden r_amplitudes = [] t_amplitudes = [] # Doorloop R-pieklocaties om overeenkomstige T-piekamplitudes te vinden voor r_peak in r_peaks: # Zoek de index van de T-piek (minimumwaarde) in het interval van R-piek tot R-piek + 200 monsters t_peak = np.argmin(X_test[i][r_peak:r_peak+200]) + r_peak # Voeg de R-piekamplitude en T toe -piekamplitude naar de lijsten r_amplitudes.append(X_test[i][r_peak]) t_amplitudes.append(X_test[i][t_peak]) #extracting singuliere waarden uit de r_amplitudes std_r_amp = np.std(r_amplitudes) mean_r_amp = np. mean(r_amplitudes) median_r_amp = np.median(r_amplitudes) sum_r_amp = np.sum(r_amplitudes) #extraheren van singuliere waarden uit de t_amplitudes std_t_amp = np.std(t_amplitudes) mean_t_amp = np.m ean(t_amplitudes) median_t_amp = np.median(t_amplitudes) sum_t_amp = np.sum(t_amplitudes) # Vind de tijd tussen opeenvolgende R-pieken rr_intervals = np.diff(r_peaks) # Bereken de tijdsduur van de gegevensverzameling time_duration = (len (X_test[i]) - 1) / 1000 # ervan uitgaande dat gegevens in ms zijn # Bereken de bemonsteringsfrequentie bemonsteringssnelheid = len(X_test[i]) / time_duration # Bereken hartslagduur = len(X_test[i]) / bemonsteringssnelheid hartslag = (len(r_peaks) / duur) * 60 # QRS-duur qrs_duration = [] for j in range(len(r_peaks)): qrs_duration.append(r_peaks[j]-r_peaks[j-1]) #extracting singular value metrics from de qrs_duartions std_qrs = np.std(qrs_duration) mean_qrs = np.mean(qrs_duration) median_qrs = np.median(qrs_duration) sum_qrs = np.sum(qrs_duration) # De standaarddeviatie van het RR-interval extraheren std_rr = np.std( rr_intervals) mean_rr = np.mean(rr_intervals) median_rr = np.median(rr_intervals) sum_rr = np.sum(rr_intervals) # Extraheren van de standaarddeviatie van de RR -interval std = np.std(X_test[i]) # Extraheren van het gemiddelde van het RR-interval gemiddelde = np.mean(X_test[i]) # Toevoegen van de kenmerken aan de lijst X_test_fe.append([mean, std, std_qrs , mean_qrs,median_qrs, sum_qrs, std_r_amp, mean_r_amp, median_r_amp, sum_r_amp, std_t_amp, mean_t_amp, median_t_amp, sum_t_amp, sum_rr, std_rr, mean_rr,median_rr,heart_rate]) # De lijst converteren naar een numpy array X_test_fe = np.array(X_test_fe)
De vorm van de testset na kenmerkextractie is als volgt:
(1000, 19)
Stap 7: Modelbouw en training
We gaan nu een LSTM-model van een terugkerend neuraal netwerk bouwen. Eerst zullen we de gegevens opnieuw vormgeven om ze compatibel te maken met het model. Vervolgens maken we een LSTM-model met slechts 2 lagen. Vervolgens trainen we het op de functies die uit de gegevens zijn gehaald. Als laatste doen we de voorspellingen op de validatie/testset.
# Definieer het aantal features in het dataframe van de trein num_features = features.shape[1] # Hervorm de feature-gegevens zodat ze de juiste vorm hebben voor LSTM-invoer features = np.asarray(features).astype('float32') features = features .reshape(features.shape[0], features.shape[1], 1) X_test_fe = X_test_fe.reshape(X_test_fe.shape[0], X_test_fe.shape[1], 1) # Definieer het model architectuurmodel = Sequential( ) model.add(LSTM(64, input_shape=(features.shape[1], 1))) model.add(Dense(1, activation='sigmoid')) # Compileer het model model.compile(optimizer='adam ', loss='binary_crossentropy', metrics=['accuracy']) # Train de modelgeschiedenis = model.fit(features, y_train, validation_data=(X_test_fe, y_test), epochs=50, batch_size=32) # Doe voorspellingen op de validatieset y_pred = model.predict(X_test_fe) # Converteer de voorspelde waarden naar binaire labels y_pred = [1 als p>0.5 anders 0 voor p in y_pred] X_test_fe = np.asarray(X_test_fe).astype('float32')
Stap 8: Modelevaluatie
Nu gaan we de prestaties van het model evalueren met behulp van metrische gegevens, bijvoorbeeld nauwkeurigheid, precisie van de AUC-score, enz. Verder gaan we de verwarringsmatrix in Plotly visualiseren.
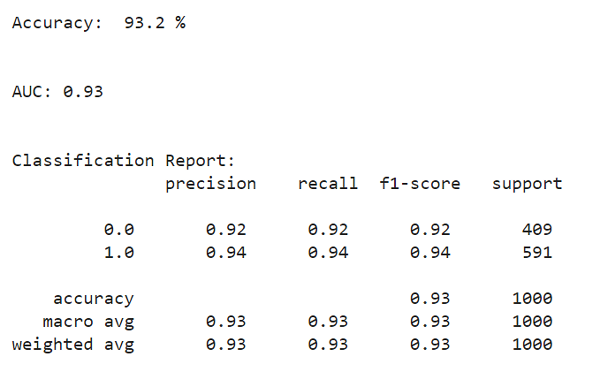
1. Alle statistieken berekenen
# bereken de nauwkeurigheid acc = accuratesse_score(y_test, y_pred) #bereken de AUC-score auc = round(roc_auc_score(y_test, y_pred),2) #classificatierapport biedt alle statistieken, bijv. precisie, terugroepactie, enz. all_met = classificatie_rapport(y_test, y_pred )
2. Alle statistieken weergeven
# Print de nauwkeurigheid print("Nauwkeurigheid: ", acc*100, "%") print(" n") print("AUC:", auc) print(" n") print("Classificatierapport: n", all_met ) afdrukken("n")
Output:
3. Berekenen en weergeven van de verwarringsmatrix
# Bereken de verwarringsmatrix conf_mat = verwarring_matrix(y_test, y_pred) conf_mat_df = pd.DataFrame(conf_mat, columns=['Predicted Negative', 'Predicted Positive'], index=['Actual Negative', 'Actual Positive']) fig = px.imshow(conf_mat_df, text_auto= True, color_continuous_scale='Blues') fig.update_xaxes(side='top', title_text='Predicted') fig.update_yaxes(title_text='Actual') fig.show()
Output:

Stap 9: de trainings- en validatiefout uitzetten
Laten we ten slotte de trainingsfout en validatiefout visualiseren na elke periode van de modeltraining.
# Plot training en validatiefout fig = go.Figuur() fig.add_trace(go.Scatter( y=history.history['loss'], mode='lines', name='Training')) fig.add_trace(go .Scatter( y=history.history['val_loss'], mode='lines', name='Validation')) fig.update_layout(xaxis_title="Epoch", yaxis_title="Error", title= {'text': 'Modelfout', 'xanchor': 'center', 'yanchor': 'top', 'x':0.5} , bargap=0) fig.show()
Output:

Resultaten
Het model behaalde een recall-waarde van 0.92 en een AUC-score van 0.93, wat zijn effectiviteit aantoont met een eenvoudige deep-learning-architectuur. In de gezondheidszorg is terugroepen een cruciale maatstaf, vooral bij het screenen van ziekten, waar fout-negatieven ernstige gevolgen kunnen hebben, waaronder gemiste kansen voor vroege opsporing en behandeling. De goede recall-score in dit project benadrukt het potentieel van dit model om de inspanningen op het gebied van ziektescreening positief te beïnvloeden.
Future Work
De gegevens zijn onevenwichtig met ongeveer 2919 voorbeelden van normale gevallen en 2079 voorbeelden van afwijkende gevallen. Het zou beter zijn om eerst de dataset in evenwicht te brengen om de Recall- en AUC-scores te verbeteren. Ook zou in de toekomst een ingewikkelder deep learning-model met meer lagen kunnen worden toegepast om de resultaten te verbeteren.
Conclusie
In dit project hebben we een uitgebreide tijdreeksgegevensanalyse en functie-extractieproces doorlopen. We hebben de gegevens gefilterd met behulp van verschillende technieken en de beste geselecteerd op basis van onze specifieke use case. Verder hebben we kenmerken uit de ECG-signalen gehaald en deze kenmerken gebruikt om een tweelaags LSTM-model te trainen. We hebben de prestaties van het model beoordeeld met behulp van metrische gegevens zoals nauwkeurigheid, AUC-score, precisie, enz., en de resultaten gevisualiseerd met behulp van een verwarringsmatrix en trainings- en validatiefoutgrafieken.
Dit zijn de belangrijkste leerdoelen van het project:
- Het belang begrijpen van het filteren van tijdreeksgegevens en het kiezen van het beste filteralgoritme.
- Praktijkervaring met kenmerkextractie uit tijdreeksgegevens, met name ECG-signalen, en de impact van deze kenmerken op de prestaties van het model.
- Het proces begrijpen van het bouwen, trainen en evalueren van een deep learning-model, met name een LSTM-model, voor afwijkingsdetectie.
- Over het geheel genomen benadrukt dit project het belang van vakkennis en feature-engineering bij het bouwen van effectieve modellen voor tijdreeksgegevens.
U heeft toegang tot de volledige jupyter notebook hier op deze link.
Bedankt voor het lezen van dit artikel! Als je de inhoud over anomaliedetectie met behulp van deep learning leuk vond en op de hoogte wilt blijven van vergelijkbare onderwerpen, volg me dan op LinkedIn.
De in dit artikel getoonde media zijn geen eigendom van Analytics Vidhya en worden naar goeddunken van de auteur gebruikt.
Verwant
- Door SEO aangedreven content en PR-distributie. Word vandaag nog versterkt.
- Platoblockchain. Web3 Metaverse Intelligentie. Kennis versterkt. Toegang hier.
- Bron: https://www.analyticsvidhya.com/blog/2023/02/anomaly-detection-in-ecg-signals-identifying-abnormal-heart-patterns-using-deep-learning/
- 1
- 10
- 28
- 39
- 7
- 9
- a
- vermogen
- Over
- boven
- toegang
- nauwkeurigheid
- bereikt
- verwerven
- Adam
- Daarnaast
- Na
- vooruit
- Steun
- wil
- algoritme
- algoritmen
- Alles
- Het toestaan
- bedragen
- analyse
- analytics
- Analytics Vidhya
- analyseren
- het analyseren van
- en
- onregelmatigheidsdetectie
- toegepast
- architectuur
- rond
- reeks
- dit artikel
- geëvalueerd
- helpen
- attributen
- vermijd
- Balance
- BAND
- gebaseerde
- omdat
- wezen
- onder
- BEST
- Betere
- tussen
- blogathon
- bouw
- Gebouw
- berekenen
- het berekenen van
- vangen
- verzorging
- geval
- gevallen
- Centreren
- kenmerken
- controle
- controleren
- Kies
- het kiezen van
- uitgekozen
- vorderingen
- classificatie
- classificeren
- code
- codering
- Collectie
- Kolom
- columns
- vergelijken
- vergelijking
- verenigbaar
- Voltooid
- het invullen van
- complex
- uitgebreid
- Berekenen
- conclusie
- verwarring
- opeenvolgend
- Gevolgen
- bouw
- content
- verband
- samentrekking
- converteren
- Overeenkomend
- kon
- bedekt
- en je merk te creëren
- Wij creëren
- cruciaal
- gegevens
- gegevensanalyse
- datasets
- deep
- diepgaand leren
- Vraag
- beschrijven
- Opsporing
- Bepalen
- afwijking
- diagnostiseren
- verschil
- anders
- direct
- Onthul Nu
- goeddunken
- Ziekte
- ziekten
- aandoeningen
- Display
- weergeven
- Verdeeld
- e-commerce
- elk
- Vroeg
- effectief
- effectiviteit
- doeltreffend
- inspanningen
- geeft je de mogelijkheid
- elimineren
- benadrukte
- Engineering
- Geheel
- Milieu
- tijdperk
- fout
- vooral
- essentieel
- etc
- Ether (ETH)
- schatten
- evalueren
- evaluatie
- EVENTS
- Alle
- voorbeeld
- voorbeelden
- duur
- ervaring
- Nagegaan
- uitdrukkelijk
- extract
- extractie
- Kenmerk
- Voordelen
- voeden
- Vijg
- Figuur
- filter
- filtering
- Tot slot
- VIND DE PLEK DIE PERFECT VOOR JOU IS
- het vinden van
- Voornaam*
- Vlotter
- volgen
- gevolgd
- volgend
- volgt
- formaat
- frauduleus
- oppompen van
- vol
- geheel
- verder
- Bovendien
- toekomst
- Go
- goed
- diagram
- grafieken
- sterk
- Gezondheid
- gezondheidszorg
- gezondheidszorg
- Hart
- Hoogte
- hulp
- helpt
- hier
- Hoge
- highlights
- geschiedenis
- houden
- Hoe
- HTTPS
- identificeren
- het identificeren van
- Imaging
- onbalans
- Impact
- Effecten
- importeren
- belang
- belangrijk
- verbeteren
- verbetert
- het verbeteren van
- in
- omvat
- Inclusief
- index
- -industrie
- informatie
- eerste
- invoer
- Introductie
- gaat
- problemen
- IT
- Keras
- kennis
- label
- labels
- Achternaam*
- Legkippen
- leiden
- LEARN
- leren
- Lengte
- niveaus
- bibliotheken
- Bibliotheek
- lijnen
- LINK
- Lijst
- lijsten
- het laden
- locaties
- uit
- Laag
- machine
- machine learning
- Hoofd
- maken
- maken
- Marketing
- Matrix
- Materie
- zinvolle
- Media
- medisch
- medische beeldvorming
- Geheugen
- methode
- methoden
- metriek
- Metriek
- minimum
- vermist
- model
- modellen
- monitor
- meer
- meest
- MS
- naam
- nav
- noodzakelijk
- Noodzaak
- negatief
- negatief
- netwerk
- Neural
- neuraal netwerk
- New
- volgende
- Geluid
- een
- notitieboekje
- aantal
- numpy
- doelstellingen
- EEN
- Kansen
- origineel
- Overige
- Overig
- geschetst
- totaal
- eigendom
- stootkussen
- panda's
- parameters
- deel
- deelnemers
- bijzonder
- patiënt
- patiëntenzorg
- patronen
- prestatie
- Plato
- Plato gegevensintelligentie
- PlatoData
- dan
- punt
- punten
- positief
- potentieel
- PRAKTISCH
- praktijk
- precisie
- voorspeld
- Voorspellingen
- Voorbereiden
- vereisten
- aanwezigheid
- probleem
- project
- projecten
- zorgen voor
- biedt
- gepubliceerde
- doeleinden
- Python
- tarief
- Rauw
- Lezer
- lezing
- verminderen
- Gereduceerd
- relevante
- het verwijderen van
- verslag
- vertegenwoordigen
- vertegenwoordigt
- REST
- resultaat
- Resultaten
- terugkeer
- RIJ
- dezelfde
- Wetenschap
- doorlichting
- Ontdek
- sectie
- sector
- Sectoren
- te zien
- gekozen
- selecteren
- -Series
- ernstig
- reeks
- Sets
- verscheidene
- Vorm
- Ploegen
- getoond
- Signaal
- signalen
- gelijk
- Eenvoudig
- vereenvoudigen
- single
- enkelvoud
- Soft /Pastel
- specifiek
- specifiek
- spleet
- Spot
- squared
- standaard
- begin
- Statement
- statistisch
- blijven
- Stap voor
- Stappen
- shop
- opgeslagen
- gestructureerde
- onderwerpen
- dergelijk
- Pieken
- neemt
- doelwit
- technieken
- tensorflow
- proef
- Testen
- De
- De toekomst
- De grafiek
- hun
- spullen
- drie
- drempel
- Door
- niet de tijd of
- Tijdreeksen
- naar
- tools
- top
- onderwerpen
- Trainen
- Trainingen
- Transacties
- Reizend
- behandeling
- Trends
- waar
- types
- begrip
- unieke
- ongebruikelijk
- bijgewerkt
- Gebruik
- .
- use case
- bevestiging
- waarde
- Values
- variëteit
- visualisatie
- visualiseren
- Voltage
- Wave
- golven
- welke
- wil
- zou
- X
- Your
- zephyrnet